科普驿站 第三十七期
主题:核酸的剪刀——CRISPR-Cas系统
科目:生物
难度:B2
讲师:杜瑾鸿
CRISPR/Cas9:出道即巅峰
1953年,詹姆斯·杜威·沃森(James Dewey Watson,1928-)与弗朗西斯·哈里·康普顿·克里克(Francis Harry Compton Crick,1916-2004)提出了DNA的双螺旋结构,标志了分子生物学的诞生;1963年,约书亚·莱德伯格提出了在人体内引入基因的概念,并认为“这仅仅是个时间问题”;1978年,因发现“发现限制性内切酶及其在分子遗传学方面的应用”,沃纳·亚伯、丹尼斯·那森斯、汉密尔顿·史密斯分享了当年的诺贝尔生理学或医学奖。
基因工程(genetic engineering)并不是一个新鲜概念,甚至基因编辑(genome editing)也不是。在本文的主角——CRISPR/Cas9登场之前,我们就能在一定程度上修改基因。随着时间推移一些,还曾有过两种基因编辑的工具(并至今有应用),即锌指核酸酶(ZFN)和转录激活因子样效应物核酸酶(TALEN),但因为商业和技术上的种种原因,它们的效率并不高。如果你读过其他关于CRISPR/Cas9的科普,就会知道Cas9是一种能够剪断DNA的酶。ZFN和TALEN同CRISPR/Cas9的一个本质区别是,前者依靠蛋白质与特定DNA结合来识别特定的序列;而CRISPR/Cas9靠的是一条RNA。正如我们从细菌中发现限制酶那样,CRISPR/Cas系统也自细菌中发现。
发现史
了解它的历史,能帮助我们理解它。1987年,石野良纯(石野 良純,Ishino Yoshizumi)等人在研究大肠杆菌的一个基因(iap)时,无意间发现了一段紧接着这个基因的“无法确定功能”的DNA(如图所示)。研究者注意到这段DNA同时具有如下特征,同时具有所有这些特征的DNA是以前的研究中从未发现的:
引自Ishino Y , Shinagawa H , Makino K , et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product.[J]. Journal of Bacteriology, 1987, 169(12):5429-5433.
1.这段DNA中的一些小片段具有几乎相同的碱基排列方式(其中一条链是CGGTTTATCCCCGCTRRCGCGGGGAACTC)。但它们并不首尾相接,而是被另外一些排列方式多变的DNA隔开,就好像是准备好很多条相同的小段,然后把它们均匀地插入了一段杂乱排列的DNA一样。这些序列称为重复序列(repeat),而把它们隔开的杂乱排列的DNA称为间隔序列(spacer)。
2.一段重复序列有29个碱基,而将它们隔开的间隔序列虽然杂乱多变,但每一段一定有32个碱基。
3.一段重复序列中有部分片段是反向互补的。比如在上图中,被下划线的部分——TCCCCGC和GCGGGGA,写出前者的反向排列(即CGCCCCT),发现它可以与GCGGGGA恰好互补,反之亦然。
这种DNA并没有立即得名,或者说只是因为类似的结构在很多细菌中被发现,而随便取了几个名字(如DR:direct repeat;TREP:tandem repeat;SRSR:short regularly spaced repeats)。然而,1989年,中田敦夫(中田 敦夫,Atsuo Nakata)、牧野公三(マキノ 公三,Kozo Makino)等人在另外两种品系的大肠杆菌,以及另外两种细菌——痢疾志贺氏菌(Shigella dysenteriae)、肠志贺氏菌(S. enterica)中再次发现了相同特征的DNA片段;1991年,彼得·赫曼斯(Peter W.M.Hermans)等人在结核分歧杆菌(Mycobacterium tuberculosis)等放线菌门(phylum Actinobacteria)成员中又发现了此类DNA;1993年,这样的DNA首次发现在古细菌——地中海嗜盐菌(Haloferax mediterranei)中。随着时间推移,科学家在越来越多的原核物种中发现了这样的DNA。到2000年,科学家确定这种序列在40%左右的真细菌和90%左右的古细菌中的存在。而且还发现在不同的细菌中,乃至在同一种细菌的不同CRISPR中,人们发现重复序列和间隔序列的排列方式、重复序列重复出现的次数都可以表现出很大差异。
真细菌界与古细菌界:把真细菌等同于真菌是一个误区。20世纪最伟大的生物学家之一——Carl Woese就细菌中发现的两类已涉及到根本性的差别——如转录因子、rRNA、tRNA的类别差异——的类群提出,应该将细菌进一步分为真细菌(Eubactria)界和古细菌(Archeobacteria或者Archaea)界。古细菌经常生活在一些极端环境,如高温、高盐的环境中,这让一些人不禁猜测古细菌可能是最原始且占优势的生命体形式。
为此,2002年,吕德·詹森(Ruud Jansen)将具有以上特征的DNA片段重新命名为CRISPR。也就是说,CRISPR,也即clustered regularly interspaced short palindromic repeats——“成簇规律性间隔短回文重复序列”,是DNA。
不过,科学家依旧迟迟无法确定它的作用,这让科学家更加好奇了。
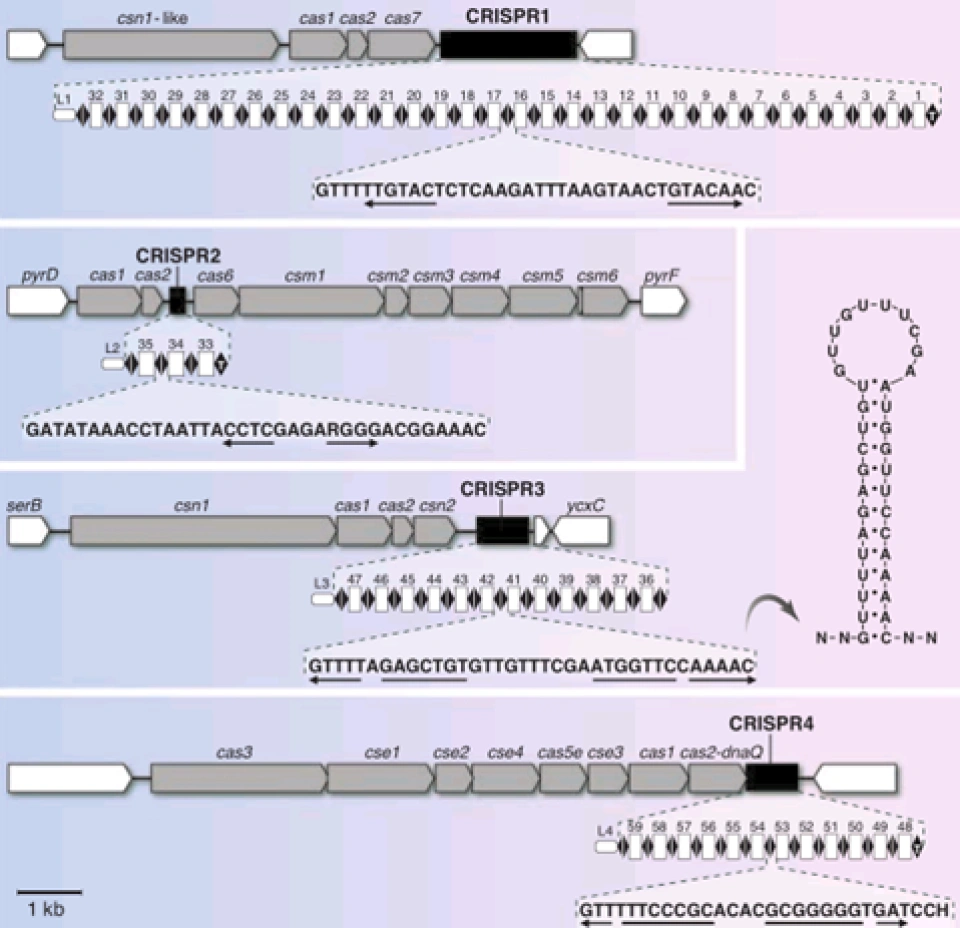
与此同时,随着测序技术的发展,科学家逐渐获得了更多细菌的基因组数据。通过比较大批细菌的基因文库,人们发现了总位于CRISPR附近的一些基因,在不同的菌种之间普遍相似。科学家最初鉴定出了4种这样的基因。这些基因被詹森命名为CRISPR-associated sequence即Cas。这4种基因分别称为Cas1-4。詹森指出:在大多数含有这4种基因的细菌中,人们发现它们在基因组中的排列顺序是Cas3-Cas4-Cas1-Cas2,换言之就是一个操纵子,CRISPR则位于这个“基因簇”的某一侧。此外,对于掘越氏热球菌(Pyrococcus horikoshii)、多杀性巴氏杆菌(Pasteurella multocida)、酿脓链球菌(Streptococcus pyogenes)等含有两个或多个CRISPR的细菌来说,在相同的细菌中,不同的CRISPR可能伴随有排列方式不同的Cas基因簇,比如下图所示的细菌。图示取自嗜热链球菌(Streptococcus thermophilus),这是一种乳酸菌。
引自Horvath P , Barrangou R . CRISPR/Cas, the Immune System of Bacteria and Archaea[J]. Science, 2010, 327(5962):167-170.
同时,在2002年的同一篇论文里,詹森还指出,Cas1和Cas2蛋白的功能无法确定,而Cas3具有解旋酶的活性;Cas4的功能则似乎与核酸外切酶相似。
这样的发现,能给我们推断它的功能带来什么用处呢?
起初人们根据Cas蛋白的功能猜测CRISPR/Cas构成了一个DNA修复系统,或者跟代谢、重组、抗高温等有关。不过有意思的是,2005年,弗朗西斯科·莫吉卡(Francisco J.M.Mojica)和克里斯汀·波塞尔(Christine Pourcel)分别独立发现间隔序列的碱基排列方式与一些噬菌体或质粒的DNA片段几乎完全相同,引发了“CRISPR与抵抗外来因子有关”的猜测,他们的研究指出,噬菌体和质粒似乎无法侵染在CRISPR有一段DNA,其排列方式与它们的某段DNA相同的细菌。除了猜测CRISPR的抵抗作用以外,他们也同时认为一定存在某种机制能够使CRISPR将入侵的DNA的关键信息记录下来。同年,亚历山大·波罗金(Alexander Bolotin)同时发现记录的噬菌体的信息越多,对噬菌体的抵抗能力就越强。已经可以说是有大量研究表明,CRISPR/Cas的存在跟细菌的获得性免疫有关。但是这些研究仍然没有引起广泛重视。2006年,基拉·马卡洛娃(Kira S.Makarova)提出并强调Cas蛋白其实是细菌获得性免疫的效应物,并提出:CRISPR/Cas“与脊椎动物的获得性免疫极为相似”。(他而且地根据CRISPR/Cas在古细菌中更加普遍的存在略为激进地推断,这种系统进化上很可能在古细菌中率先产生,然后才通过细菌间遗传物质的共享转移到了真细菌中。)
外切酶与内切酶的区别:外切酶是能够从多聚核苷酸链的末端开始水解核酸的核酸酶;内切酶是能够从多聚核苷酸链的中间开始水解核酸的核酸酶。
2007年,鲁道夫·巴兰戈(Rodolphe Barrangou)用嗜热链球菌做了一系列实验:在它的CRISPR的间隔序列中插入一些某种噬菌体的DNA,结果发现嗜热链球菌对这种噬菌体产生抵抗性;删除噬菌体基因组中,排列方式与细菌的间隔序列相同的DNA,结果发现细菌对该噬菌体失去免疫能力;这样证实了CRISPR发挥获得性免疫的作用的猜想。他们还发现CRISPR也能干扰质粒的转移。次年,约翰·范德奥斯特(John van der Oost)又证实了,CRISPR可以转录出一些特殊的RNA,这些RNA与Cas蛋白质结合成“核酸-蛋白复合体”,以此来发挥功能。实验证据和观测证据在这段时间里越来越多。最终,人们从而确切地知道了CRISPR/Cas系统。再后来,人们陆续发现了CRISPR/Cas9作用的完整的分子生物学机制,从而发现并挖掘了它改造为基因编辑工具的巨大潜力。
延伸阅读:History of CRISPR-Cas from Encounter with a Mysterious Repeated Sequence to Genome Editing Technology,这篇文章的作者之一是CRISPR的发现者之一石野良纯。本表根据该文及鲁道夫·巴兰戈与约翰·范德奥斯特所编CRISPR-Cas Systems:RNA-Mediated Adaptive Immunity in Bacteria and Archaea、鲁凤民等《CRISPR/Cas9介导的基因编辑技术》、周诺等《CRISPR/Cas9基因编辑系统的发展及其在医学研究领域的应用》及诸文献汇编而来。
我们下期继续介绍。
参考资料:
Haft D H , Selengut J , Mongodin E F , et al. A Guild of 45 CRISPR-Associated (Cas) Protein Families and Multiple CRISPR/Cas Subtypes Exist in Prokaryotic Genomes[J]. PLoS Computational Biology, 2005, 1(6):e60.
Makarova K S , Haft D H , Barrangou R , et al. Evolution and classification of the CRISPR–Cas systems[J]. Nature Reviews Microbiology, 2011, 9(6):467-477.
在2005年的研究中,科学家首次为Cas蛋白做了分类,将45种蛋白归入鉴定出的10个亚型:核心蛋白(core proteins)、Ecoli亚型、Ypest亚型、Nmeni亚型、Dvulg亚型、Tneap亚型、Hmari亚型、Apern亚型、Mtube亚型、RAMP组件一共十个家族中。
Ecoli亚型(cse)来源于一个大肠杆菌品系:Escherichia coli K12-MG1655。
Ypest亚型(csy)来源于多个鼠疫耶尔森菌耶尔森菌(Yersinia pestis)品系。
Nmeni亚型(csn)来源于一个脑膜炎奈瑟氏球菌品系:Neisseria meningitidis Serogroup A Z2491。
Dvulg亚型(csd)来源于希尔登堡脱硫弧菌(Desulfovibrio vulgaris Hildenborough)。
Tneap亚型(cst)来源于一个新阿波罗栖热袍菌品系:Thermotoga neapolitana DSM4359。
Hmari亚型(csh)来源于一个死海盐盒菌品系:Haloarcula marismortui ATCC 43049。
Apern亚型(csa)来源于一个敏捷气热菌品系:Aeropyrum pernix K1。
Mtube亚型(csm)来源于两个结核分枝杆菌品系:Mycobacterium tuberculosis CDC1551和H37Rv。
RAMP组件(RAMP module)亚型(cmr)是一个特殊的Cas蛋白家族。
虽然在2011年进行了重新分类,但是很多蛋白根据该分类方法所赋予的名称代号却被沿用了下来,但也有很多蛋白的名字被更改。如Cas9曾被称作Csn1或Csx12。更多详细的资料可参见TIGRFAMs(http://www.jcvi.org/cgi-bin/tigrfams/index.cgi)。
【本文为耀星会的原创作品,未经允许,禁止盗用、转载、篡改文章,否则耀星会将追究版权责任。】






![[s-70]](https://www.bokeyuan.net/pic/image/emoji/cas/70.png)












