科普驿站 第三十三期
主题:粘合素与DNA环
难度:B2
讲师:杜瑾鸿
人的一个细胞里的DNA就长达2米,细胞究竟是凭借何种高超的技术将DNA缠起来,变成尺寸只有几微米的染色体的呢?
这么长的DNA放在这么小的细胞核里,究竟是如何做到不打结呢?
细胞有时候需要让DNA链上相隔很多个碱基的DNA片段的两个片段相互靠近以促进基因表达,这是怎么实现的呢?
当你翻阅教材时,你注意到了或尝试思考过这些小细节吗?而这一切的秘密,可能都隐藏在一种叫做粘合素(cohesin)的蛋白质身上。
粘合素似乎并没有什么特殊的化学功能,例如催化或者传递化学信号,又或者特异性结合DNA来完成什么事情。可是它的功能依然极为重要,以至于在正常人体内检测到它失活突变的概率非常低,它的氨基酸排列方式在人和老鼠之间都很相似。但它只是一个朴素的环状蛋白质,可它又似乎不是一个环状肽:
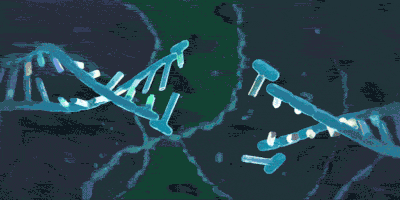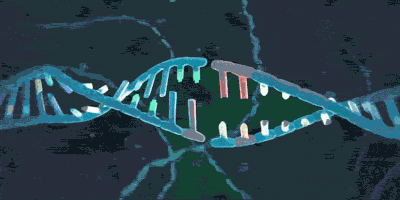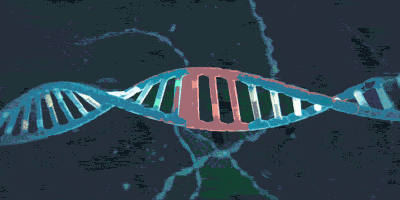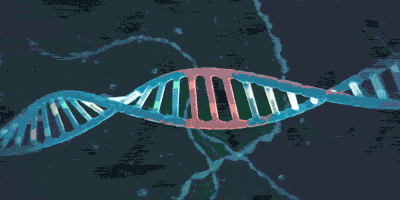
它可以套住DNA:
或者:
但却不用大费周章地从末端像穿针一样将DNA套进环内,因为它也有着四级结构——它是由多个蛋白质组成,从而来一同发挥功能的。在需要时,这几个蛋白质之间的结合可以被解除,这样就能拆开环,把DNA套进来或者从DNA上把环给拿掉。
它包含着四个组分——这部分比较晦涩,也许读者可以选择性阅读:
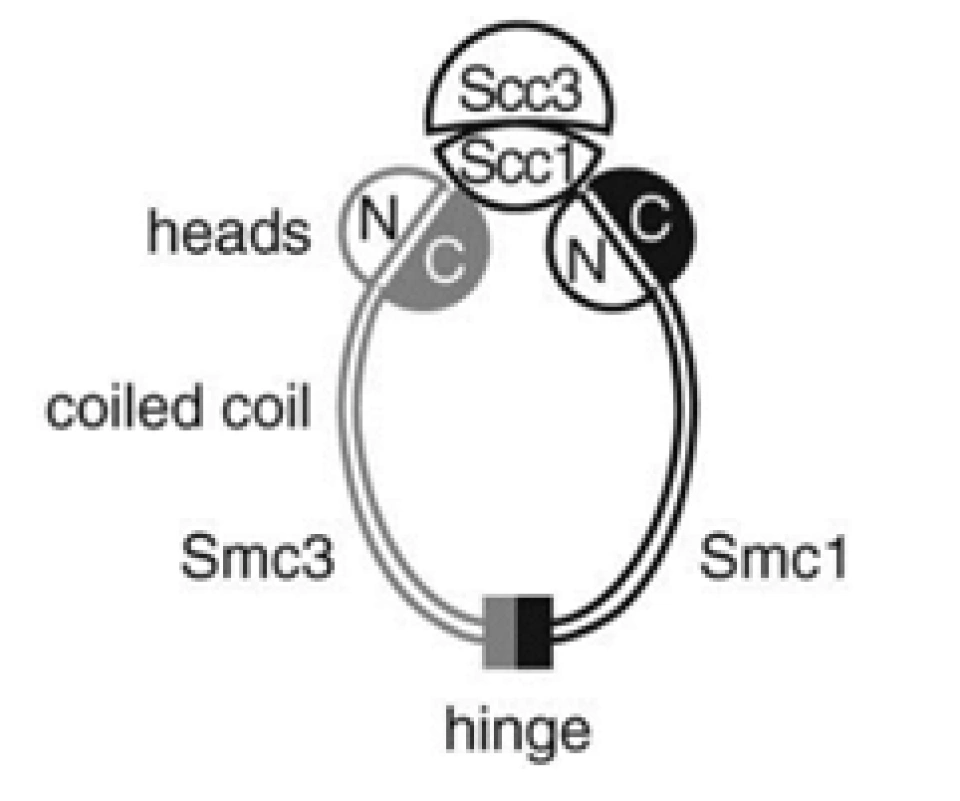
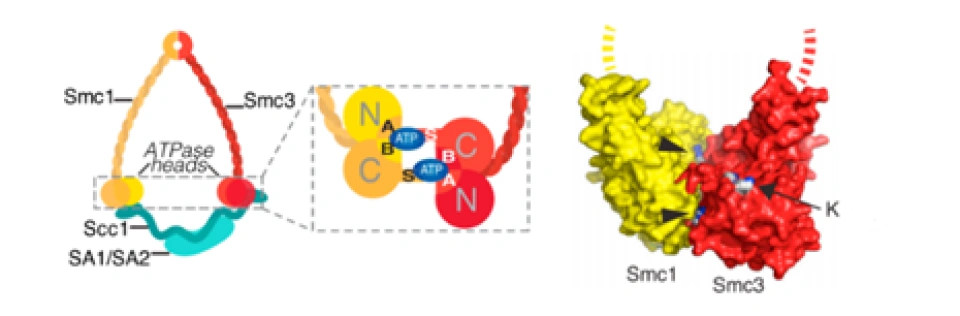
染色体结构维持蛋白(structural maintenance of chromosome protein, SMC):包括SMC1与SMC3这两种蛋白质。SMC1又分A、B两种,但只有其中的某一种构成了粘合素环。在有丝分裂时,SMC1A参与粘合素的构成;在减数分裂时,则是SMC1B。
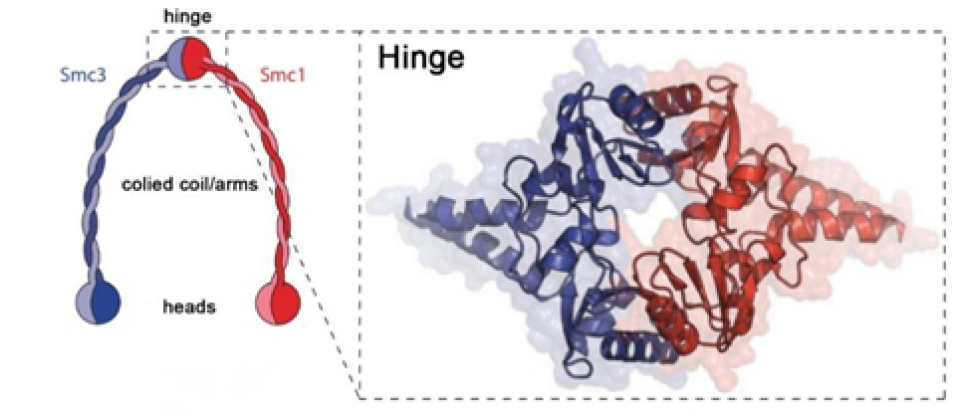
染色体结构维持蛋白构成了粘合素的主体部分,即它的卷曲螺旋(coiled coil)(或者说臂(arm))与合叶(hinge)。它们的肽链首先卷曲起来,然后被对折。这样重合的两条肽链再相互卷曲缠绕,形成像DNA那样的双螺旋结构。它们被对折的地方被经过了一定的处理,使它们俩可以通过这个部分相互结合成为合叶(hinge):
这样,就使它们形成了一个“Λ”形蛋白质。科学家们长期都在争论,到底是合叶部分,还是“头”(heads)的部分,成为了粘合素将DNA套在环中和从环中释放出来的缺口,而最近的研究发现,这两种方法可能都会使用。
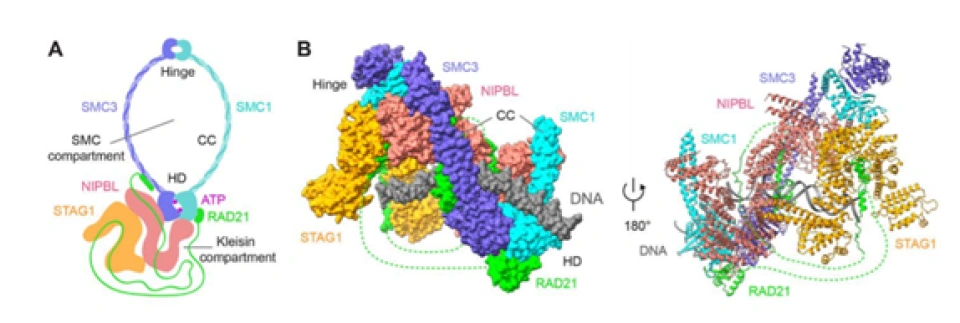
SMC1和SMC3的肽链的两端在头部形成了一种特殊的空间结构,叫做核苷酸结合结构域(nucleotide-binding domain, NBD),而且它还具有水解ATP的活性。正是依靠这种结构,SMC1-SMC3在头部(heads)通过ATP的相互结合,构成了粘合素的“内门”(inner gate)。在下图中,A、B、S是蛋白质的三种不同的空间结构。(它们其实是有全称的,A和B的全称是“脚架”A(walker A)和脚架B,S是信号(signature)的首字母。)正是它们通过与ATP结合,从而关闭了“内门”。
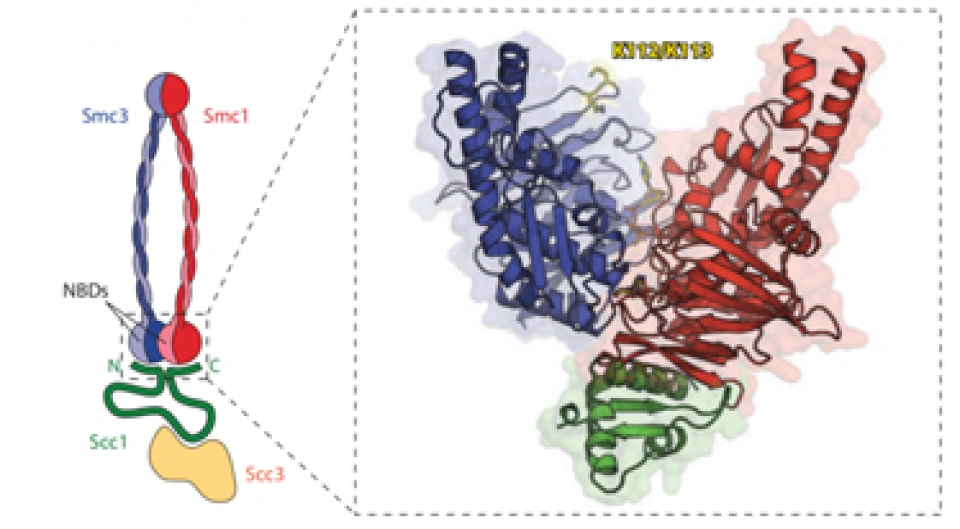
α-封闭蛋白(kleisin):这个蛋白质的中文名作者暂时没有见到中文文献中出现,这里姑且译成“封闭蛋白”,因为Kleise或Κλεισε是希腊语“闭嘴”“关闭”的意思,“封闭”一词也比较符合它的功能特点。封闭蛋白是一个大家族,除α之外,还包括β-封闭蛋白、γ-封闭蛋白等类别。对于粘合素来说,它需要用到的α-封闭蛋白可能是——
Rad21(有丝分裂时);
重组蛋白REC8(REC取自recombination的前三个字母)(减数分裂时);
Rad21L(Rad21-like,就是“跟Rad21很相似”的意思);
非SMC粘合蛋白I亚单位H(non-SMC condensin I complex subunit H, NCAPH)——
这四种蛋白质中的一种或几种。具体来说,到底取哪种蛋白质,依细胞内的不同场合与不同时间而有所不同。因为它们含有一个翼螺旋结构域(winged-helix domain, WHD),所以又叫WHD亚单位。α-封闭蛋白与SMC1-SMC3的头部(heads)结合起来,就形成了粘合素的“外门”(outer gate),要将DNA套进来,必须把内门和外门都打开才行。
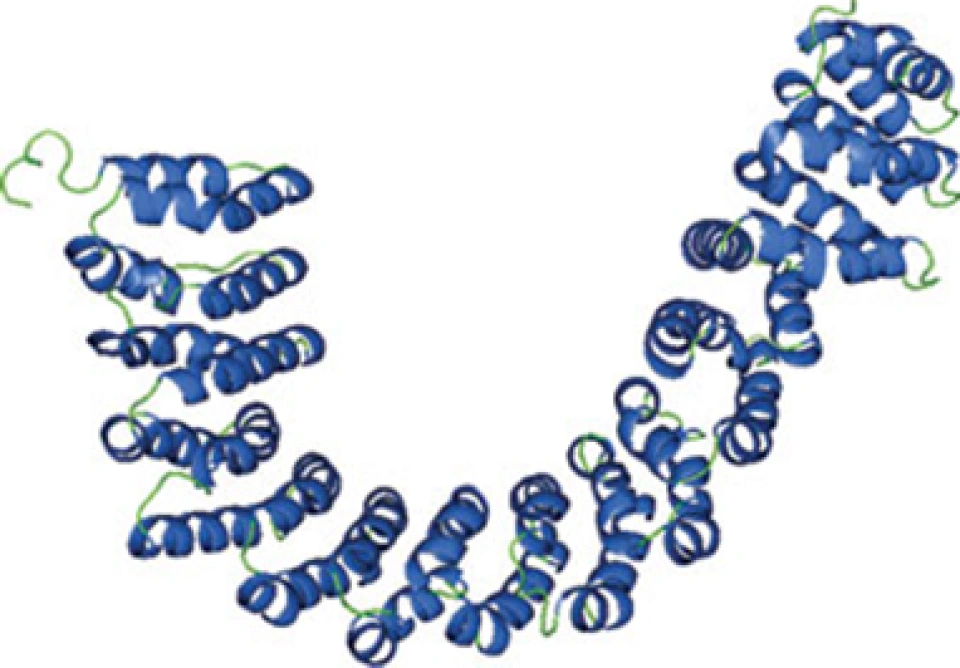
HEAT重复序列亚单位:包括基质抗原1(stromal antigen 1, SA-1或者STAG1, Scc1)、SA-2(STAG2, Scc3)以及SA-3(SA-1与SA-2出现在有丝分裂时,往往同时出现;SA-3出现在减数分裂时)。HEAT这个名称来源于三种蛋白质——亨廷顿病细胞延伸因子3(Huntington elongation factor 3,HEF3)、PR65/A(PR是protein phosphatase即蛋白磷酸酶的缩写)、TOR(雷帕霉素靶蛋白(target of rapamycin)的缩写)。下图展示了蛋白磷酸酶PR65/A的HEAT重复序列:
“重复序列”,顾名思义,就是相同的氨基酸排列方式(连同空间结构一起)在一条肽链(一个蛋白质)上重复出现了很多次——在上图中可以看到,HEAT序列重复出现了15次左右。
HEAT重复序列相关亚单位:包括姐妹染色单体早期成熟分离蛋白5(precocious dissociation of sisters protein 5,Pds5,或称姐妹染色单体粘连蛋白pds5(sister chromatid cohesion protein pds5))的两个同源体蛋白质——Pds5A和Pds5B(又叫Aprin)。
HEAT重复序列亚单位和HEAT重复序列相关亚单位到底扮演者什么样的角色呢?也许你能从下文中获取一些信息。
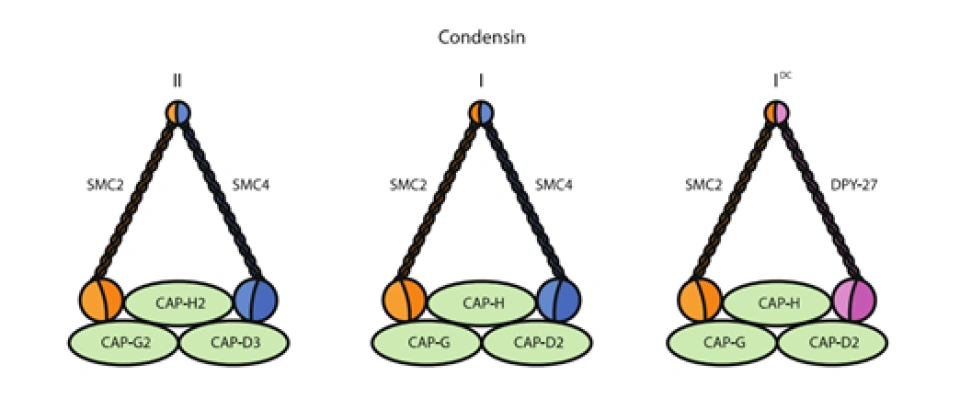
粘合素还有很多近亲,例如粘连蛋白(condensin):
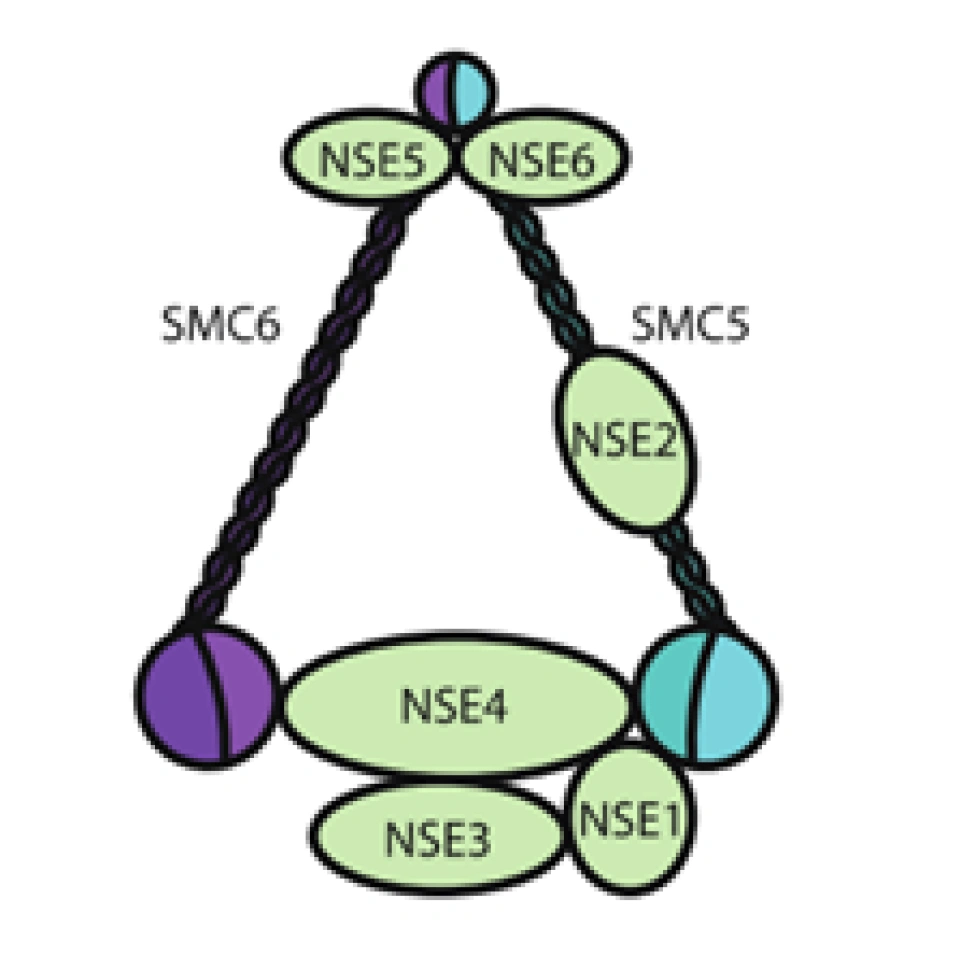
SMC5/6:
实际上,粘合素套住DNA的机制,因这个过程的后续作用(或者它们参与的生命活动,比如有丝分裂或者减数分裂,再或者基因表达调控)的不同而变化。这里只是粗略地加以介绍。
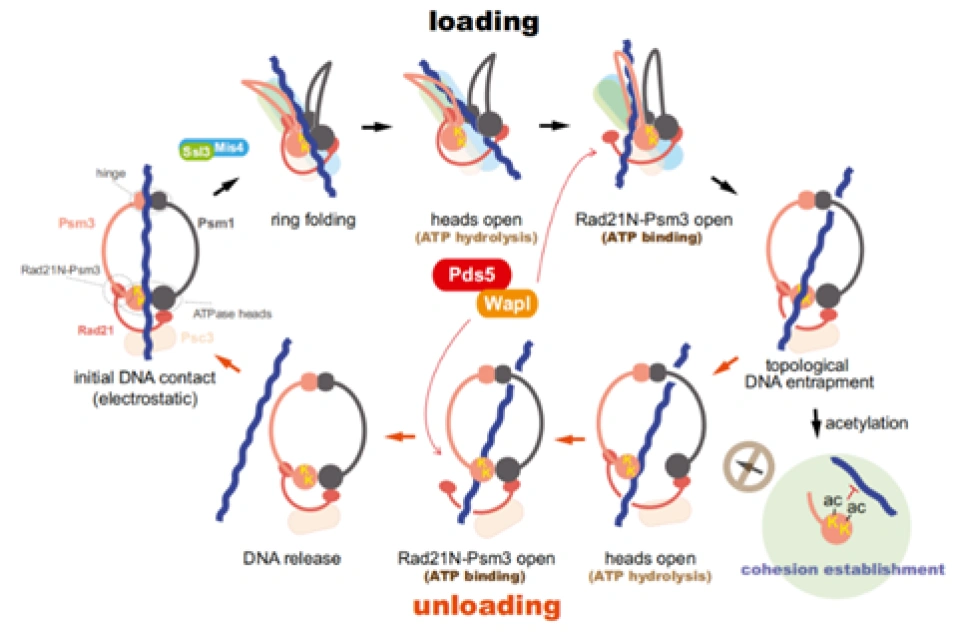
在套环的过程开始之前,粘合素复合体的头(heads)与合叶(hinge),会与DNA通过静电吸引相互靠近(上图的“initial DNA contact”步骤)。然后,一个由两种特殊的蛋白质——Scc2(又叫Nipped-B-like protein(NIPBL)或者Mis4)和Scc4(又叫Ssl3)(Scc家族的另外两个成员——SA-1(Scc1)和SA-2(Scc3))——组成的异源二聚体蛋白,与SMC1-SMC3的合叶(hinge)部分相结合,导致SMC1和SMC3从中间再次被对折(上图中的“ring folding”步骤)。
Scc2-Scc4这个复合体称作黏结蛋白(kollerin),有时又被称作“加载器”(loader),它事先通过结合DNA上的其他蛋白质从而被间接锚定在DNA上。Kollerin这个复合蛋白,最初在2004年被美国科学家戴尔·多赛特(Dale Dorsett)称为黏附素(adherin)——因为adhere这个动词意思就是“黏着/附着”。后来,在2011年,一位英国科学家,也是粘合素方面的研究专家——基姆·内史密斯(Kim Nasmyth)提议改为“kollerin”——这个词的词源是希腊语的Κολλαο即“用胶水粘附”的意思。内史密斯认为adherin这个词容易与钙黏蛋白的cadherin混淆,且也没有很好地概括它的功能。同样地,这个蛋白质的中文名作者暂时没有见到中文文献中出现,这里译成了“黏结蛋白”。研究发现黏结蛋白可能是通过与其他蛋白质相互作用,从而被固定在染色质丝上的,换言之,有其他的蛋白质事先与DNA结合,黏结蛋白与它结合,才得以被锚定在DNA上。这个“其他蛋白质”可能是下列蛋白质中的一种或几种——
细胞分裂周期蛋白7(cell division cycle 7,cdc7)-DBF4相关因子1(Dbf4-related protein 1, drf1)激酶(DDK);
中心体蛋白P(centromere protein P, CENPP,又叫ctf19);
CENPN(又叫染色体丢失蛋白4(chromosome loss protein 4,CHL4)、染色体传递保真度蛋白(Chromosome transmission fidelity protein 17,CTFP17)、小染色体维持蛋白17(minichromosome maintenance protein 17,MMP17));
CENPL(又叫小染色体丢失增长蛋白3(increased minichromosome loss protein 3,Iml3)、MMP19);
哑铃形成蛋白4(dumbbell forming protein 4, Dbf4,又叫DDK激酶调节亚单位DBF4(DDK kinase regulatory subunit DBF4)
但是科学家并不是那么确定。
对折之后,SMC1-SMC3的头部水解ATP。ATP是连接SMC1和SMC3的两个核苷酸结合结构域的桥梁,没有了ATP,SMC1-SMC3复合体的头部,也就是内门被打开了(上图的“heads open”步骤)。与此同时,SMC1-SMC3同时招募一个由Pds5和半翼蛋白(wings apart-like protein, Wapl)组成的异源二聚体(简称Pds5-Wapl),催化Rad21与SMC3之间的结合被拆开,这样一来粘合素环外门就打开,粘合素环因此也就被完全打开了(上图的“Rad21N-Psm3 open”步骤)。然后,粘合素环恢复自己的形状,从而将DNA套进来。然后Rad21与SMC3重新结合,SMC3的肽链的112号和113号位置的两个赖氨酸(赖氨酸的缩写是K。它的R基上有一个氨基,因为在除羧基以外的第5个碳原子,所以称作ε-氨基(ε是第五个希腊字母))的氨基被两种N-乙酰化酶——Esco1与Esco2的其中一种各加上一个乙酰基(Ac),导致它不能靠近DNA,这个过程的完成,被称为粘合的建立(cohesion establishment)。这个乙酰基无法被去掉。
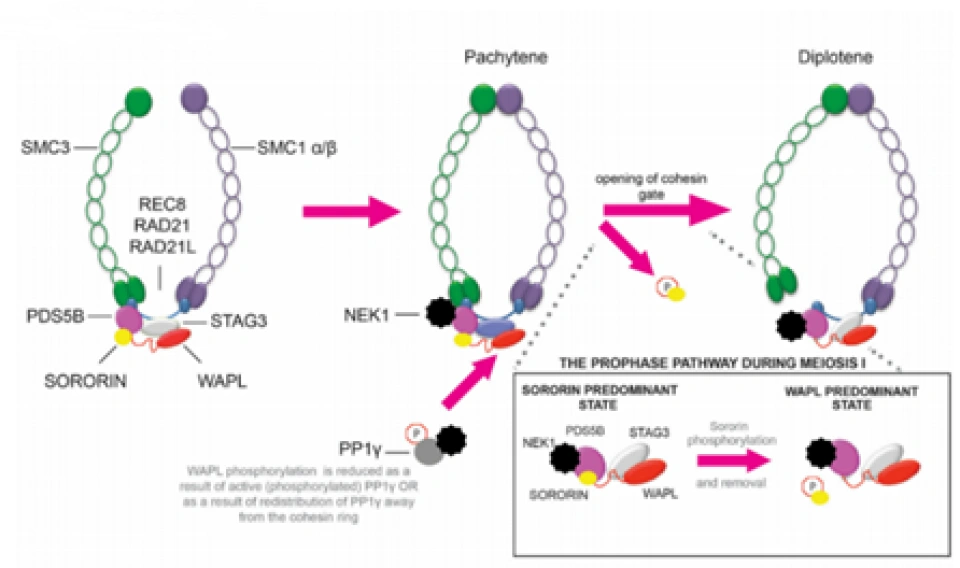
这两个乙酰基发挥的作用有时不仅是防止DNA靠近它。在细胞周期G1期到了后期阶段的时候,与SMC1-SMC3的头部结合了pds5-scc3与α-封闭蛋白,由于头部被两个乙酰基锁死而无法打开。于是乎,黏结蛋白可能会强行拆开SMC1-SMC3的合叶(hinge),从而将环套在DNA上。在减数分裂时,情况则又不同,由Pds5B、SA-3、Wapl,以及另外一个蛋白质——sororin结合在α-封闭蛋白上。(sororin又叫细胞分裂周期相关蛋白5(Cell division cycle-associated protein 5,CDCA5),sororin这个名字取自拉丁语soror,即姐妹的意思。所以sororin应该可以译成“姐妹蛋白”。)在粘合素的一旁,哺乳动物NIMA样激酶1(Mammalian NIMA-like kinase-1,NEK1)(NIMA是never in mitosis gene A的缩写,NIMA是一个蛋白质)结合含有一个磷酸基的蛋白磷酸酶1γ(PP1γ),将姐妹蛋白磷酸化,导致姐妹蛋白脱离粘合素,并使NEK1与Pds5B结合,催化α-封闭蛋白与SMC3的连接断开,同样也能够完成DNA的套环。
粘合素的卸载(unloading)并不是套环的反演,因为被乙酰化的粘合素的头部是无法被打开的。并且它的机制也更加多变了。有一个机制是在Wapl、姐妹蛋白、Pds5B在场的情况下,招募分离酶(separase),将scc1切开,然后将DNA从环中释放出来。或者,对于SMC3头部没有乙酰化的粘合素来说,则是在头部水解它结合的两个ATP的作用下将内门打开,然后Pds5-Wapl被重新招募过来,并同时结合肌醇六磷酸(IP6)(肌醇就是环己六醇,IP6就是它的六个羟基全都加上磷酸基。)催化封闭蛋白与SMC3的连接断开,也就打开了外门,从而将DNA释放出来。
总之,粘合素的套环和卸载过程都比较复杂,还涉及到各种各样的调节因子,所以我们很难做准确的定性描述。
我们下期介绍。
Key publications & References
[1] Nasmyth, Kim. Cohesin: a catenase with separate entry and exit gates?[J]. Nature Cell Biology, 2011, 13(10):1170-1177.
[2] Nasmyth, K., & Haering, C. H. (2009). Cohesin: Its Roles and Mechanisms. Annual Review of Genetics, 43(1), 525–558. doi:10.1146/annurev-genet-102108-134233
[3] Murayama Y , Samora C P , Kurokawa Y , et al. Establishment of DNA-DNA Interactions by the Cohesin Ring[J]. Cell, 2018, 172(3):465-477.e15.
[4] Chao, W. C. H., Murayama, Y., Muñoz, S., Costa, A., Uhlmann, F., & Singleton, M. R. (2015). Structural Studies Reveal the Functional Modularity of the Scc2-Scc4 Cohesin Loader. Cell Reports, 12(5), 719–725. doi:10.1016/j.celrep.2015.06.071
[5] Murayama Y , Uhlmann F . DNA Entry into and Exit out of the Cohesin Ring by an Interlocking Gate Mechanism[J]. Cell, 2015, 163(7):1628-1640.
[6] Ladurner, R., Bhaskara, V., Huis in ’t Veld, P. J., Davidson, I. F., Kreidl, E., Petzold, G., & Peters, J.-M. (2014). Cohesin’s ATPase Activity Couples Cohesin Loading onto DNA with Smc3 Acetylation. Current Biology, 24(19), 2228–2237. doi:10.1016/j.cub.2014.08.011
[7] Weitzer, S., Lehane, C., & Uhlmann, F. (2003). A Model for ATP Hydrolysis-Dependent Binding of Cohesin to DNA. Current Biology, 13(22), 1930–1940. doi:10.1016/j.cub.2003.10.030
[8] Livak, F., & Nussenzweig, A. (2019). One ring to rule them all. Nature. doi:10.1038/d41586-019-03200-4
[9] Chan, K.-L., Roig, M. B., Hu, B., Beckouët, F., Metson, J., & Nasmyth, K. (2012). Cohesin’s DNA Exit Gate Is Distinct from Its Entrance Gate and Is Regulated by Acetylation. Cell, 150(5), 961–974. doi:10.1016/j.cell.2012.07.028
[10] Sanborn, A. L., Rao, S. S. P., Huang, S.-C., Durand, N. C., Huntley, M. H., Jewett, A. I., … Aiden, E. L. (2015). Chromatin extrusion explains key features of loop and domain formation in wild-type and engineered genomes. Proceedings of the National Academy of Sciences, 112(47), E6456–E6465. doi:10.1073/pnas.1518552112
[11] Aragon, L., Martinez-Perez, E., & Merkenschlager, M. (2013). Condensin, cohesin and the control of chromatin states. Current Opinion in Genetics & Development, 23(2), 204–211. doi:10.1016/j.gde.2012.11.004
[12] Aaron F Severson, Barbara J Meyer. Divergent kleisin subunits of cohesin specify mechanisms to tether and release meiotic chromosomes[J]. Elife, 2014, 3(35):e03467.
[13] Susannah Rankin (2005) Sororin, the Cell Cycle and Sister Chromatid Cohesion, Cell Cycle, 4:8, 1039-1042, DOI: 10.4161/cc.4.8.1926
[14] Noia D , Javier M . Molecular biology: Unequal opportunity during class switching[J]. Nature, 2015.
[15] Shi Z , Gao H , Bai X C , et al. Cryo-EM structure of the human cohesin-NIPBL-DNA complex[J]. Science, 2020:eabb0981.
[16] Brieño-Enríquez, M. A., Moak, S. L., Toledo, M., Filter, J. J., Gray, S., Barbero, J. L., … Holloway, J. K. (2016). Cohesin Removal along the Chromosome Arms during the First Meiotic Division Depends on a NEK1-PP1γ-WAPL Axis in the Mouse. Cell Reports, 17(4), 977–986. doi:10.1016/j.celrep.2016.09.059
[17] Ganji, M., Shaltiel, I. A., Bisht, S., Kim, E., Kalichava, A., Haering, C. H., & Dekker, C. (2018). Real-time imaging of DNA loop extrusion by condensin. Science, 360(6384), 102–105. doi:10.1126/science.aar7831
[18] Phillips, J. E., & Corces, V. G. (2009). CTCF: Master Weaver of the Genome. Cell, 137(7), 1194–1211. doi:10.1016/j.cell.2009.06.001
Figure origins
第1面上:Ref.[7]
第1面中:维基百科+作者PS
第1面下:Ref.[15]
第2面上:Ref.[2]+作者PS
第2面中:Ref.[6]
第2面下:Ref.[2]+作者PS
第3面上:https://www.cst-c.com.cn/contents/resources-protein-domains-interactions/heat-protein-domain/domains-heat
第3面下:Ref.[11]
第4面上:Ibid.
第4面下:Ref.[5]
第5面:Ref.[16]
第6面上:Ref.[17]
第6面下:Ref.[1]
第7面上:Ref.[18]
第7面下:Ref.[10]
第8面上:《环球科学》2019.4 p49+作者PS
第8面中:Dorsett, D., & Merkenschlager, M. (2013). Cohesin at active genes: a unifying theme for cohesin and gene expression from model organisms to humans. Current Opinion in Cell Biology, 25(3), 327–333. doi:10.1016/j.ceb.2013.02.003
第8面下:Ref.[3]
第9面(1):Waldman T . Emerging themes in cohesin cancer biology[J]. Nature Reviews Cancer, 2020.
第9面(2):Ref.[12]
第9面(3):Acquaviva, L., Boekhout, M., Karasu, M.E. et al. Ensuring meiotic DNA break formation in the mouse pseudoautosomal region. Nature (2020). https://doi.org/10.1038/s41586-020-2327-4
第9面(4):Ref.[8]
第9面(5):Ref.[14]
【本文为耀星会的原创作品,未经允许,禁止盗用、转载、篡改文章,否则耀星会将追究版权责任。】



![[s-70]](https://www.bokeyuan.net/pic/image/emoji/cas/70.png)